在生物工程领域,基因组规模的代谢模型(GEMs)被广泛用于预测微生物的行为。然而,传统的GEMs主要考虑了化学计量约束,忽略了酶浓度、动力学参数和途径热力学对反应通量的影响,这导致了模拟预测与实验结果之间存在显著差异。为了解决这一问题,研究者们开发了酶约束模型(ecModels),这些模型通过整合酶浓度的限制,成功地提高了化学品生产的效率。
在此基础上,中国科学院天津工业生物技术研究所生物设计中心开发了一个名为ECMpy 2.0的Python工具包,用于自动化构建和分析酶约束模型。ECMpy 2.0通过自动化获取酶动力学参数和利用机器学习预测这些参数,显著提高了参数覆盖率。此外,ECMpy 2.0引入了常见的分析和可视化功能,使得计算结果更易于用户使用。更重要的是,ECMpy 2.0整合了三种已发布的算法,这些算法利用ecModels来发现代谢工程的潜在靶点,从而为代谢工程提供了新的策略。ECMpy 2.0作为一个开源工具,可通过GitHub或pip包获取,为合成生物学和系统生物学研究提供了强大的支持。
该研究得到了国家重点研发计划、国家自然科学基金、中国科学院先导专项、天津市合成生物技术创新能力提升行动等项目资助,并在Synthetic and Systems Biotechnology期刊上发表。中国科学院天津工业生物技术研究所毛志涛副研究员为论文第一作者和共同通讯作者,马红武研究员为论文的共同通讯作者。
论文链接
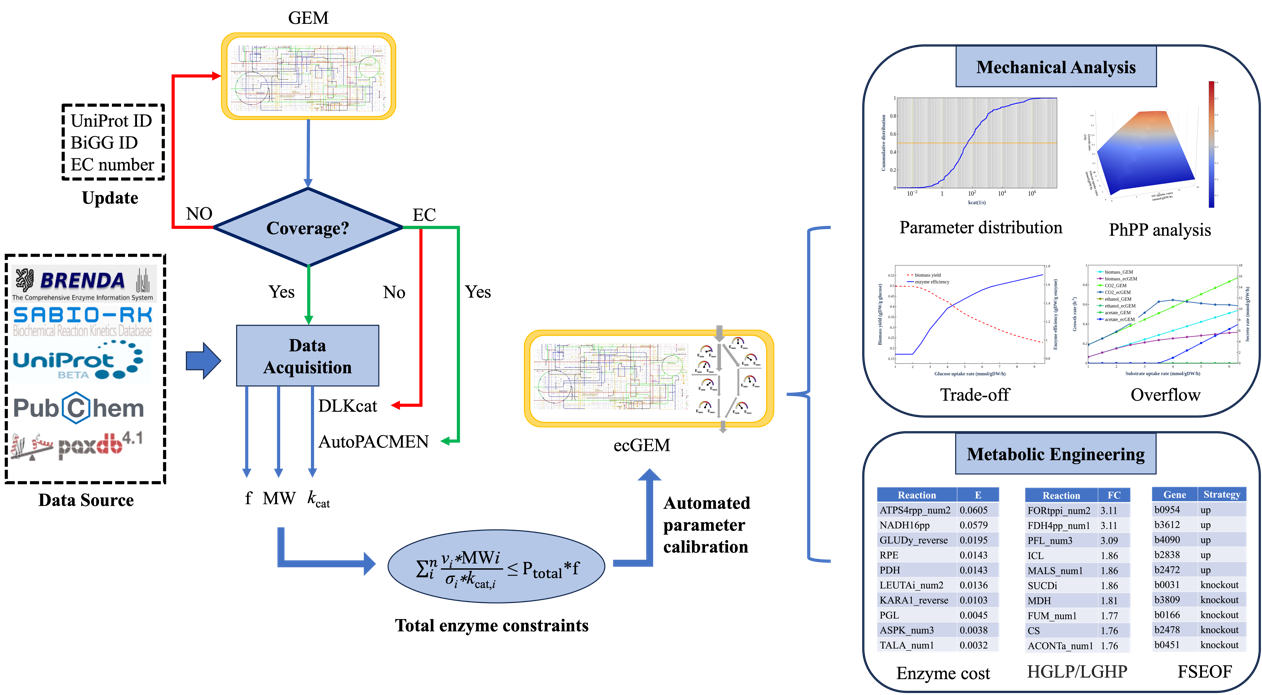
ECMpy 2.0酶约束模型构建和分析框架
