碱基编辑(base editing,BE)作为一种前沿的基因组编辑技术,能够在基因组水平上实现精确、高效的单碱基编辑。该技术已广泛应用于基础研究、基因治疗和细胞工厂构建等领域。常用的DNA碱基编辑器主要是通过将可编程的DNA结合蛋白(如Cas9)与碱基脱氨酶融合实现的,包括胞嘧啶碱基编辑器(CBE)、腺嘌呤碱基编辑器(ABE)以及糖基化酶碱基编辑器(GBE)等,可以实现C-to-T、A-to-G以及C-to-G等种类的碱基编辑。然而,这些碱基编辑器主要针对C和A碱基的直接编辑,并且它们所包含的脱氨酶可能导致非Cas9依赖的DNA或RNA脱靶。
中国科学院天津工业生物技术研究所毕昌昊研究员带领的合成生物技术研究团队和张学礼研究员带领的微生物代谢工程研究团队开发了不依赖脱氨酶(deaminase-free, DAF)的碱基编辑器DAF-CBE和DAF-TBE,分别在大肠杆菌中实现C-to-A、T-to-A的碱基颠换,在哺乳动物细胞中实现C-to-G、T-to-G的碱基颠换编辑。
研究团队首先通过定向进化改造了人源尿嘧啶糖基化酶(UNG)的两个突变体UNG (N204D)和UNG (Y147A),获得了两种高活性的DNA糖基化酶,分别可以作用于胞嘧啶碱基的CDG4和胸腺嘧啶碱基的TDG3。随后,研究团队将这两种DNA糖基化酶与nCas9(Cas9,D10A)融合,构建了CDG4-nCas9和TDG3-nCas9两种碱基编辑器,用于在大肠杆菌中进行C-to-A和T-to-A的编辑。实验结果显示,CDG4-nCas9和TDG3-nCas9在大肠杆菌中的编辑效率最高分别达到58.7%和54.3%。接着,研究团队针对Homo sapiens密码子优化版本的CDG4-nCas9和TDG3-nCas9,在HEK293T细胞中实现了C-to-G和T-to-G的颠换编辑,编辑效率分别达到38.8%和48.7%。而且,这两种编辑器的脱靶效果低于常用的胞嘧啶碱基编辑器(BE4max)和糖基化酶碱基编辑器(CGBEs)。因此,研究团队将这两个编辑器命名为DAF-CBE和DAF-TBE。此外,通过进一步的工程改造,研究团队优化了CDG和TDG的空间位置,得到了DAF-CBE2和DAF-TBE2的新版本,它们的编辑窗口从原来的间隔序列(protospacer sequence)5'端移动到中间区域,且C-to-G和T-to-G的编辑效率分别提高了3.5倍和1.2倍。最后,DAF-CBE和DAF-TBE成功实现了人诱导多功能干细胞 (hiPSC) 高效编辑。
综上所述,经过定向进化改造,开发的DAF-CBEs和DAF-TBEs碱基编辑器在大肠杆菌和哺乳动物细胞中实现了高效的碱基颠换编辑,无需使用脱氨酶。与现有的引导编辑器(prime editing)或糖基化酶碱基编辑器(GBEs)相比,DAF-BEs具有相当的编辑效率、更小的尺寸和更低的脱靶率,扩展了碱基编辑器的编辑类型,为工业菌株铸造和生物医药等领域相关研究提供了新的技术工具。
该研究工作得到了国家重点研发计划、国家自然科学基金、天津市合成生物技术创新能力提升行动、中国科学院青年创新促进会和天津市自然科学基金的支持。相关结果于近日发表在Nature Biotechnology期刊。论文第一作者为天津工业生物所博士生叶立军、副研究员赵东东以及天津师范大学生命科学学院李菊副教授。论文通讯作者为天津工业生物所张学礼研究员和毕昌昊研究员。
论文链接
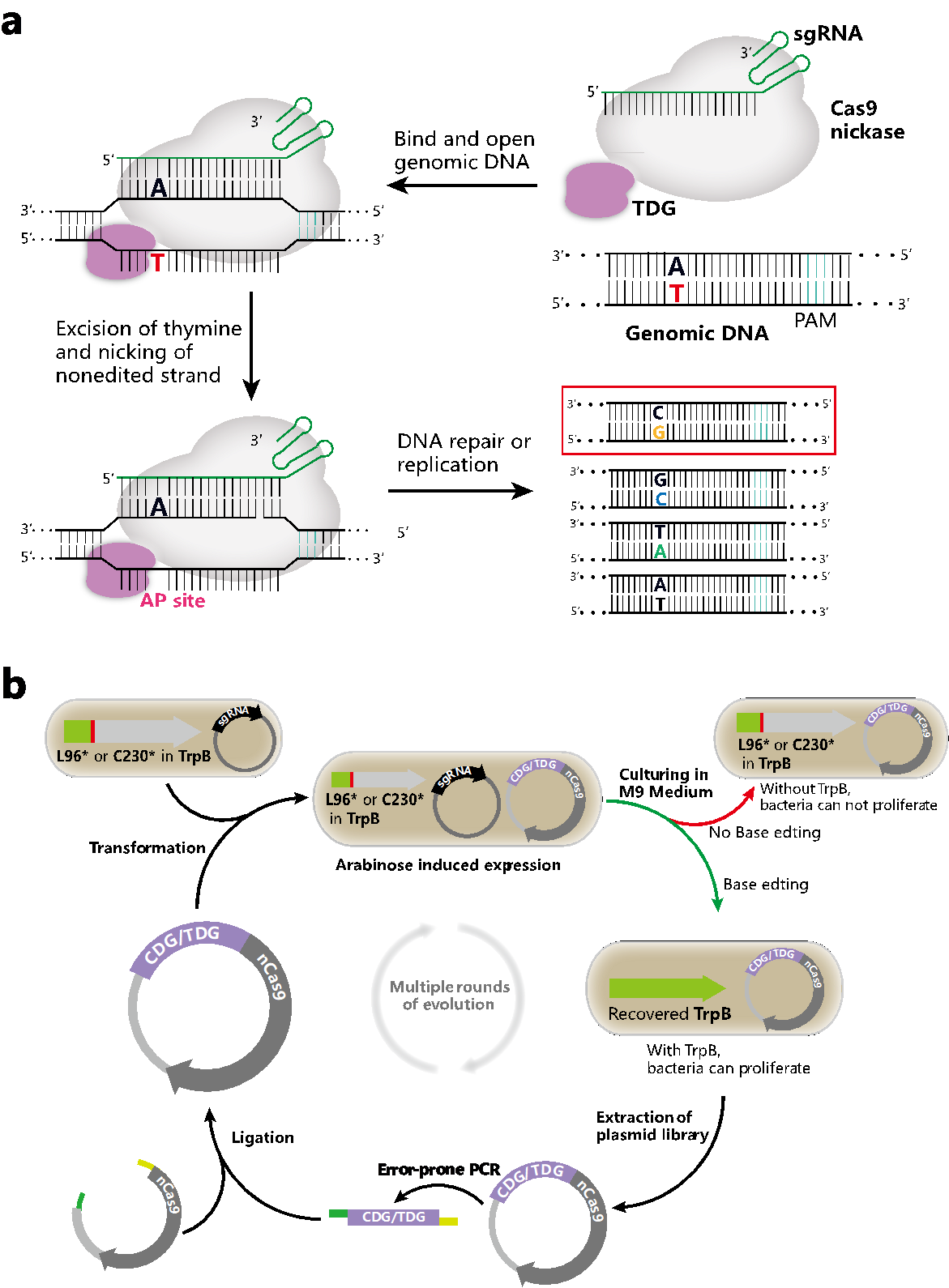
DAF-BEs碱基编辑器的设计以及进化
