单核苷酸变异(Single nucleotide variations,SNVs)是最常见的人类致病突变形式,碱基编辑技术是治疗致病性SNV的理想解决方案。NCBI的ClinVar数据库报道了超过37,000种已知的致病性SNV,其中有将近一半的致病性SNV是C>T的突变。此类突变可通过腺嘌呤碱基编辑器(Adenine base editor, ABE)进行纠正。在使用碱基编辑器纠正SNV时,如何尽量避免产生除目标核苷酸以外的任何其他核苷酸产生的旁观者编辑(Bystander editing)带来的不良影响,是碱基编辑治疗技术的关键。
近日,毕昌昊研究员带领的合成生物学技术研究团队和张学礼研究员带领的微生物代谢工程研究团队在前期igRNA(imperfect guide RNA, igRNA)编辑设计的基础上,利用慢病毒整合的方式构建了整合ABE可纠正的5000多种存在潜在旁观者编辑的SNV靶序列及相关gRNA的HeLa细胞文库,并为每个靶序列分别设计了多个igRNA。利用NG-ABEmax碱基编辑器对细胞文库进行高通量碱基编辑,收集细胞进行PCR扩增和高通量测序,对测序结果进行统计、分析,最终通过高通量筛选最佳igRNA策略,筛选出用于ABE可纠正的致病性SNV的最佳igRNA,为千种致病/可能致病SNV提供了精准治疗方案,尤其是为一些严重疾病例如罕见遗传疾病1型原发性高草酸尿症、伴有脑铁积累的神经退行性疾病等提供了高效精确的编辑策略,实现了极高的单窗口编辑效率和极少的旁观者编辑,为ABE可纠正的致病性SNV疾病的遗传疗法的开发提供了可用平台,对基因疗法的发展具有很高的价值。
相关工作得到国家自然科学基金、国家重点研发计划、天津市合成生物技术创新能力提升行动、天津市自然科学基金项目资助。相关成果发表在Molecular Therapy期刊。天津工业生物所博士研究生李博和副研究员赵东东为该论文共同第一作者,毕昌昊研究员、张学礼研究员为该论文共同通讯作者。
文章链接
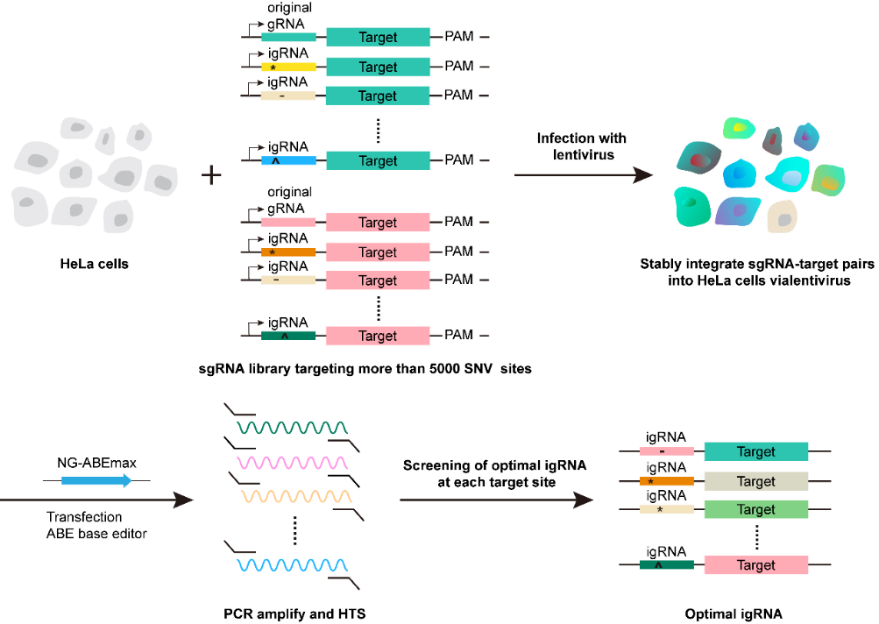
高通量筛选方法筛选ABE可纠正的致病性SNV的最佳igRNA流程图
