GBE编辑器可实现碱基C-to-G的编辑,但其仍存在碱基编辑效率偏低、靶向位点范围受限等问题,这大大限制了GBE碱基编辑器的应用。因此,亟需对现有糖基化酶碱基编辑器进行优化,开发出碱基编辑效率高、靶向位点范围广的GBE突变体。
近日,中国科学院天津工业生物技术研究所毕昌昊研究员带领的合成生物技术研究团队、张学礼研究员带领的微生物代谢工程团队在优化糖基化酶碱基编辑器方面取得新进展。为了提高GBE碱基编辑器的编辑效率,研究团队首先将转录激活因子VP64融合表达在GBE编辑器脱氨酶的氨基端(Vp64-APOBEC-nCas9-UNG),结果表明Vp64-APOBEC-nCas9-UNG的C-to-G编辑效率比GBE有显著提高。为了进一步扩大APOBEC-nCas9-UNG的应用范围,研究团队分别融合表达了SunTag系统与SpRY-Cas9,构建了SunTag-GBEs和SpRY-GBEs。与GBE相比,SunTag-GBEs在哺乳动物细胞中的编辑效率、编辑纯度和编辑框都出现了显著提升。同时SpRY-GBEs克服PAM序列的限制,实现了对靶向位点范围的拓展。该研究通过不同策略对GBE进行优化,获得性能更为优越的GBE突变体。
该研究工作得到国家自然科学基金、国家重点研发计划、天津市合成生物技术创新能力提升行动和中国博士后科学基金的资助,相关研究结果发表于Cell Reports期刊。天津工业生物所客座硕士研究生董兴啸和博士后杨超是该论文的共同第一作者,大连工业大学的陈明教授、天津工业生物所张学礼研究员和毕昌昊研究员为该论文的共同通讯作者。
文章链接
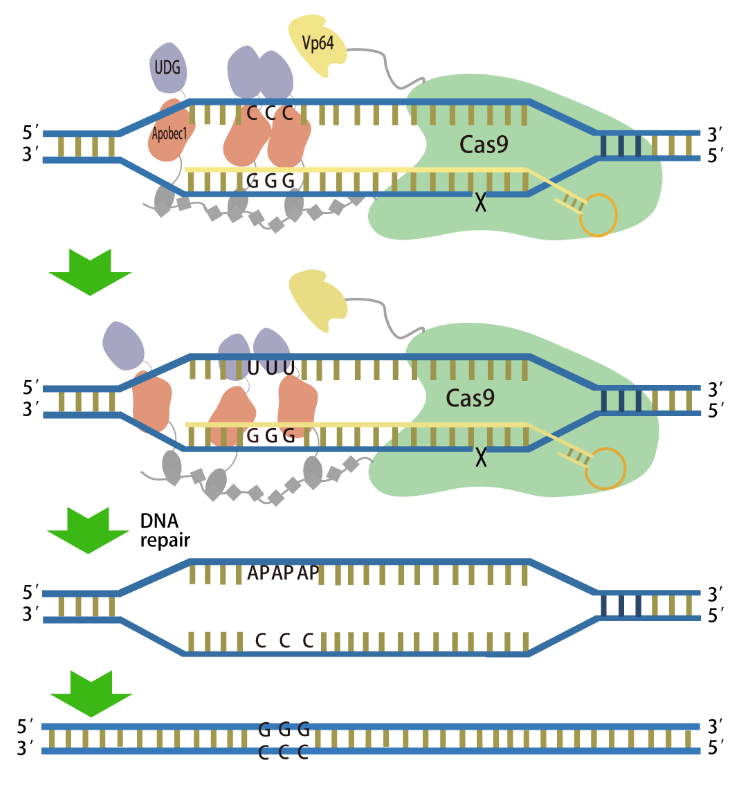
SunTag-GBEs的工作示意图
