碱基编辑技术可实现精确的碱基转换,目前有三类碱基编辑器被广泛应用,包括胞嘧啶碱基编辑器(cytosine base editor, CBE),腺嘌呤碱基编辑器(adenine base editor, ABE),以及糖基化酶碱基编辑器(glycosylase base editor, GBE)。
中国科学院天津工业生物技术研究所毕昌昊研究员带领的合成生物技术研究团队、张学礼研究员带领的微生物代谢工程团队在2020年合作研究开发GBE,可实现胞嘧啶到鸟嘌呤的碱基颠换,具有纠正3000多个已知致病单核苷酸变异(single nucleotide polymorphism,SNP)的潜力,这可能为主要的遗传疾病,如abca4相关性视网膜疾病和X-linked视网膜色素变性等带来治疗方案。
为了评估GBE在多个位点的编辑情况,提高其应用潜力,研究人员通过高通量细胞建模技术,在哺乳动物细胞中进行了6400多个基因组靶点的GBE编辑,并建立了人工智能AI机器学习模型。将收集到的数据用于AI训练,成功得到了预测碱基编辑效率和结果比例的模型。同时,基于获得的序列基序,可以设计具有高编辑效率的GBE gRNA,并且在293T和HeLa 两种细胞中证实了GBE机器学习模型可以近似预测任意给定GBE gRNA的编辑结果。此外,研究人员利用该模型预测了人类疾病相关点突变的建模和校正的编辑结果,提出一系列具有高预测编辑效率的SNP,进一步为单碱基突变引起的疾病的治疗带来了希望。这项工作克服了GBE技术的序列依赖问题,显著提高了其应用能力。
相关工作得到国家自然科学基金、国家重点研发计划、天津市合成生物技术创新能力提升行动、天津市自然科学基金项目的资助。相关成果发表在Journal of Genetics and Genomics期刊。中国科学院天津工业生物技术研究所博士生李博、博士后李娅秋和副研究员赵东东为该论文的共同第一作者,毕昌昊、张学礼为该论文的共同通讯作者。
文章链接
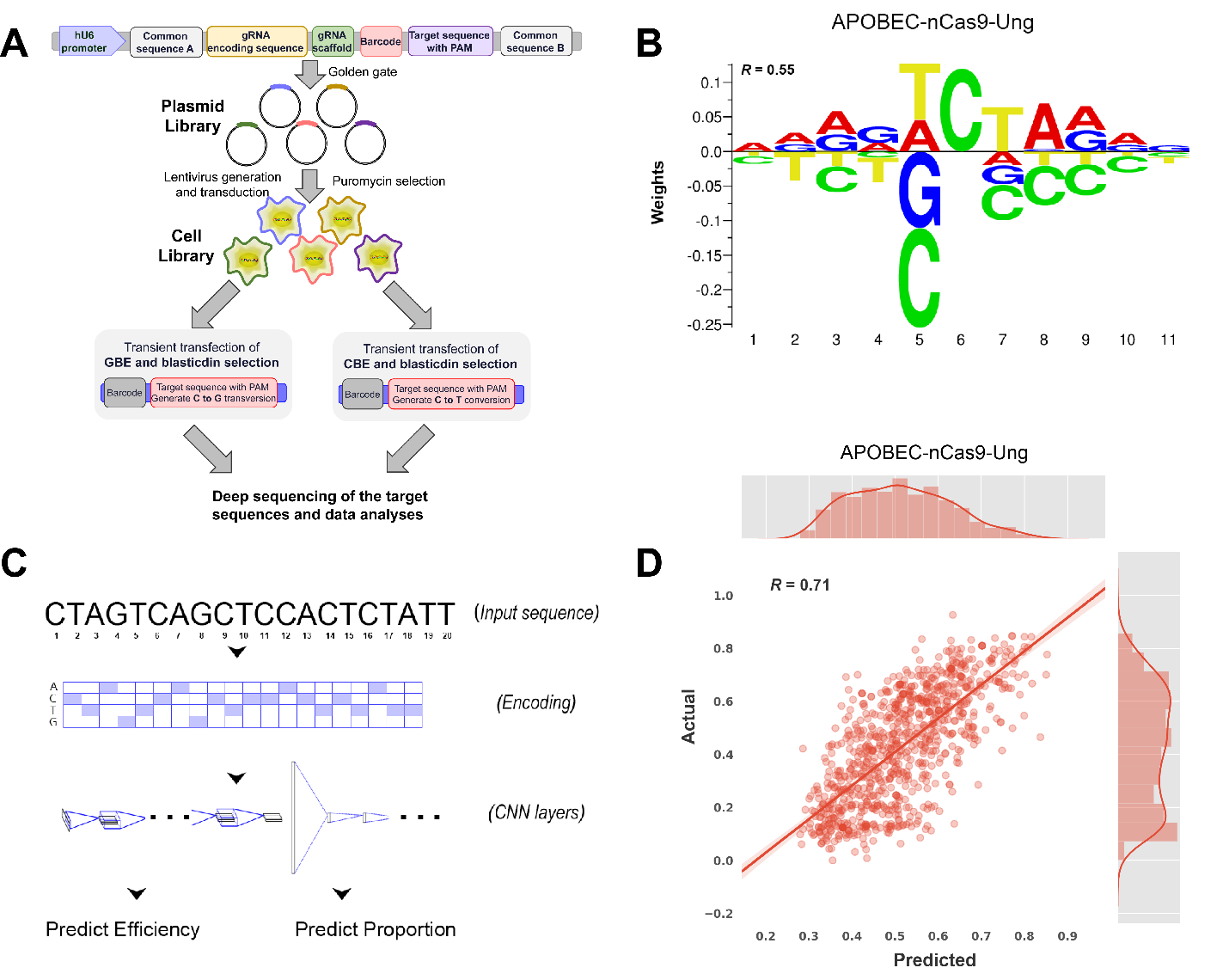
GBE编辑结果的AI机器学习模型的构建思路
