链霉菌是许多重要天然产物的生产者,其基因组蕴含着大量未被开发的次级代谢生物合成基因簇。传统的基于双链断裂的CRISPR/Cas9技术虽然已成功应用于链霉菌的基因组编辑,但需提供外源修复模板,且在多位点同时编辑的应用上仍有一定的局限性。近年来发展起来的单碱基编辑技术已成功应用于天蓝色链霉菌等一些模式菌株中,相较于传统CRISPR技术更为方便快捷。碱基编辑的效率与底盘菌株尿嘧啶DNA糖苷酶(UDG)的功能密切相关,前期研究多用枯草芽孢杆菌噬菌体来源的尿嘧啶DNA糖苷酶抑制因子UGI来提升碱基编辑效率,但在某些情况下,其抑制效果却并不理想。
中国科学院天津工业生物技术研究所王猛研究员带领的高通量编辑与筛选平台实验室,与天津科技大学花而并教授团队合作,在模式菌株变铅青链霉菌Streptomyces lividans 66中研究了UDG与碱基编辑效率的关系,并开发了新一代碱基编辑器asRNA-BE(antisense RNA interference-enhanced CRISPR/Cas9 Base Editing method)。团队首先利用融合了胞苷脱氨酶rAPOBEC1的BE系列碱基编辑器成功实现基因组三个次级代谢基因的编辑。在此基础上,利用CRISPR/Cas9辅助的基因敲除手段,分别构建了尿嘧啶DNA糖苷酶UDG1和UDG2的单敲和双敲菌株,发现在失活UDG1的情况下,碱基编辑效率可较原始提高3.4倍到67.4倍不等。考虑到UDG是细胞修复过程中的关键酶,其长期缺失不利于菌株基因组的稳定,团队利用反义RNA干扰降低基因表达水平的策略代替基因敲除,在原始的BE编辑器基础上整合了针对UDG的反义RNA干扰模块,构建了新一代碱基编辑器asRNA-BE,成功将编辑效率提高至原来的2.8倍到65.8倍不等。asRNA-BE编辑器可以在碱基编辑过程中瞬时抑制UDG的表达,而在编辑结束后又可以通过温敏型质粒的消除恢复UDG的表达水平,在提高编辑效率的同时避免了对于细胞不可逆的伤害。基因组测序结果也表明,与BE编辑器相比,asRNA-BE编辑器在大幅度提高编辑效率的同时,并未引起额外的脱靶,在工业菌株改造等方面具有良好的应用前景。
该研究得到国家重点研发计划、天津市合成生物技术创新能力提升行动、中国科学院重点项目以及国家自然科学基金的支持,相关研究结果以封面文章的形式发表在ACS Synthetic Biology上。天津工业生物技术研究所助理研究员张玥为论文的第一作者。
文章链接

封面文章
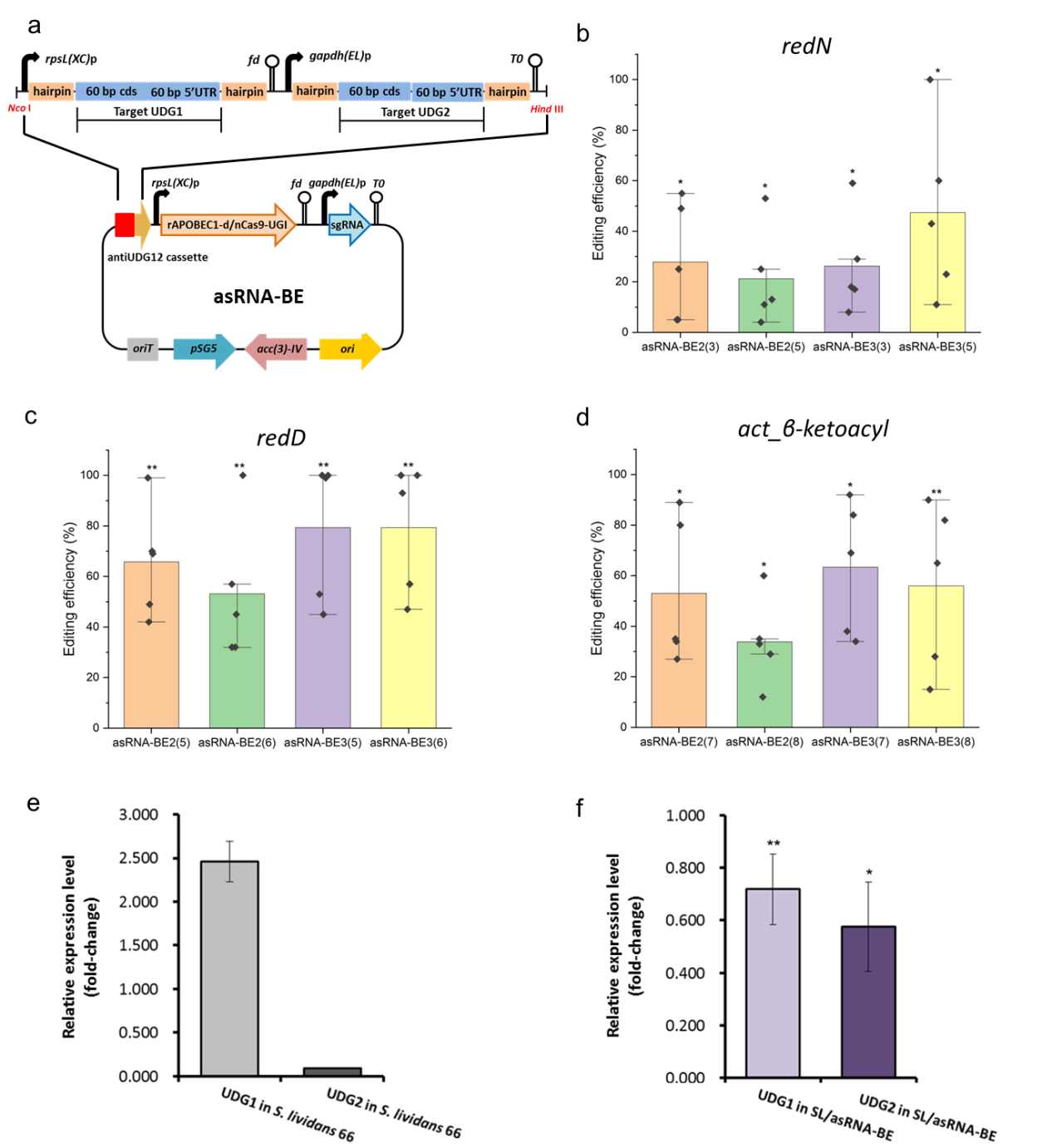
asRNA-BE编辑器在变铅青链霉菌中的应用
