由蛋白酶所调控的底物水解对于所有生命形式具有重要的基础意义。失控的蛋白酶底物水解则跟许多人类疾病密切相关,包括癌症、炎症以及心血管疾病等。蛋白酶功能研究的核心瓶颈是如何准确识别所有的天然底物。因而,掌握蛋白酶底物特异性知识能够极大提高我们预测酶底物以及设计抑制剂的能力。
中科院天津工业生物技术研究所结构生物信息学与整合系统生物学课题组围绕胱天蛋白酶(Caspases,又称半胱天冬酶)和颗粒酶B(Granzyme B)的底物裂解特异性开展了一系列深入的研究。本工作开发出最新的生物信息学工具Cascleave 2.0(http://www.structbioinfor.org/cascleave2/),用以精确识别caspase-1, 3, 6, 7, 8 和Granzyme B的酶切裂解位点。这一研究通过整合多种不同来源的蛋白质序列和结构信息来提高Cascleave 2.0的预测精确度。在特征选择过程中,本工作使用了最大相关最小冗余(mRMR)和向前特征选择(FFS)算法。与研究中测试的其他工具相比,Cascleave 2.0工具对caspase-1, 3, 6, 7 和Granzyme B达到了最优的预测效果。这一生物信息学工具的成功开发,对于快速筛选这些蛋白酶的专一性裂解靶标蛋白和深入研究蛋白酶-底物调控关系具有极其重要的意义。
该研究得到中国科学院知识创新项目(KSCX2-EW-G-8)、天津专项(10ZCKFSY05600)和国家自然科学基金(61202167)的资助。最新研究结果发表在由英国牛津大学出版社出版的、生物信息学领域的顶级杂志Bioinformatics (五年影响因子6.911)上,天津工业生物所结构生物信息学与整合系统生物学课题组科研助理王明君博士为论文第一作者。
文章链接:http://www.ncbi.nlm.nih.gov/pubmed/24149049
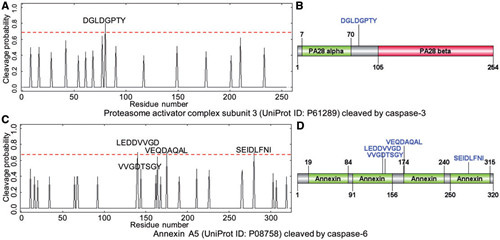
使用Cascleave 2.0预测caspase裂解位点的可能性
